COVID-19 : Comment le variant sud-africain se coupe un bras pour échapper à l’immunité

Cette équipe de biologistes de l’IBM computational biology center vient de décrypter comment la variante sud-africaine hautement infectieuse du SRAS-CoV-2, connue sous le nom de B.1.351, parvient à cet « échappement immunitaire » en cas d'infection préalable et en cas de vaccination. L’équipe décrit dans le Journal of Medicinal Chemistry comment l'une des trois mutations qui rendent le variant B.1.351 différent de la souche originale, réduit la liaison du virus aux cellules humaines dans l’objectif, d'ailleurs atteint, d'échapper à certains anticorps.
Depuis que le SARS-CoV-2 original a été détecté pour la première fois fin de 2019, plusieurs nouvelles variantes ont émergé, au Royaume-Uni, en Afrique du Sud et au Brésil. Parce que les nouveaux variants semblent plus transmissibles et se propager rapidement, de nombreuses personnes craignent qu'ils soient résistants aux vaccins actuels, aux thérapies par anticorps voire à l'immunité naturelle. Quelques études ont déjà ainsi suggéré que les vaccins actuels pourraient ne pas être suffisamment efficaces contre certains des nouveaux variants. D'autres études documentent déjà l'infection après la vaccination.
Une 3è mutation déroutante
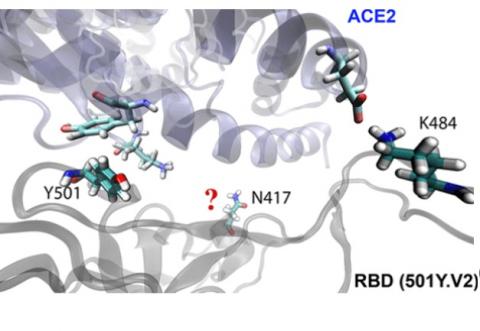
La variante B.1.351 porte 2 mutations (N501Y et E484K) qui peuvent améliorer la liaison entre le domaine de liaison au récepteur (RBD) de la protéine de pointe de coronavirus et le récepteur ACE2 humain. Cependant, la 3è mutation (K417N) semble jouer contre le virus et contre la réussite de l’infection, car elle élimine une interaction favorable entre la RBD et l'ACE2. Les chercheurs Binquan Luan et Tien Huynh d'IBM Research ont donc cherché quelle pouvait être la raison d’être ou l’avantage évolutif d'une telle mutation.
Quel avantage évolutif d'une telle mutation ? Des simulations de dynamique moléculaire ont permis aux scientifiques d’analyser les effets de la mutation K417N dans la variante B.1.351.
Les scientifiques ont d’abord modélisé la liaison entre le SRAS-CoV-2 original et l'ACE2 via le domaine de liaison RBD, et entre le RBD et le CB6, qui est un anticorps neutralisant contre le SRAS-CoV-2 : ils découvrent alors que l'acide aminé d'origine, une lysine, en position 417 dans le RBD, interagit plus fortement avec l’anticorps CB6 qu'avec le récepteur ACE2, ce qui explique au demeurant l'efficacité thérapeutique de l'anticorps chez les animaux modèles.
Ensuite, l'équipe a modélisé la liaison avec le variant à mutation K417N. La mutation induit la transformation de la lysine en un autre acide aminé, l’asparagine et cette transformation réduit la force de la liaison entre le RBD et l'ACE2 et, simultanément, de la liaison du RBD à CB6 et à plusieurs autres anticorps humains.
Ainsi, le variant B.1.351 n’hésite pas à sacrifier l’une de ses liaisons à ACE2 pour échapper à la réponse anticorps du système immunitaire.
Une nouvelle illustration de la complexité de l’évolution du virus et des données utiles pour améliorer la protection des thérapies et vaccins actuels contre les variants.
