COVID-19 : Un modèle open source de la protéine de pointe « S »
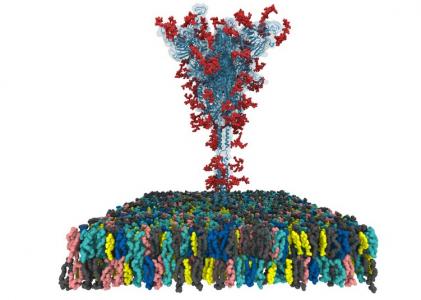
Cette équipe internationale vient de développer le tout premier modèle open source au niveau atomique de la protéine de pointe « S » ou « Spike » du coronavirus SARS-CoV-2. Voilà donc décryptée et modélisée, dans le Journal of Physical Chemistry B, la cible clé pour le développement de vaccins et de médicaments anti-COVID-19.
On sait aujourd’hui que la protéine de pointe S facilite l'entrée du virus dans les cellules hôtes ; précisément, le coronavirus utilise sa protéine de pointe pour se lier à 2 protéines de liaison présentes sur cellules humaines, ACE2 et CD147. Comprendre la structure de la protéine S et pouvoir bloquer ses interactions avec les protéines de liaison peut permettre de bloquer le processus d’infection.
Les consignes de reproduction du modèle en accès libre
Cette équipe de virologues de l'Université de Corée du Sud (Séoul) avec des collègues de l'Université de Cambridge et de la Lehigh University (Pennsylvanie) vient de produire les premiers modèles open source de la protéine S et livre avec ses travaux une vidéo qui explique la structure de ce système. Les consignes permettant de reproduire ces modèles sont en accès libre (puis cliquer sur COVID-19 Archive ).
CHARMM-GUI (GUI : graphical user interface ) est un programme qui simule les systèmes biomoléculaires complexes de manière simple, précise et rapide. Son auteur, Wonpil Im, professeur de Biologie et de Bioingénierie à la Lehigh University explique aux scientifiques que ce modèle permet de comprendre les interactions au niveau moléculaire qui ne peuvent être observées autrement.
Le résultat d’un travail de modélisation très complexe mais qui sera précieux pour la conception de traitements et d’un vaccin contre COVID-19.
